为什么归一化
将每个特征的影响“公平化”。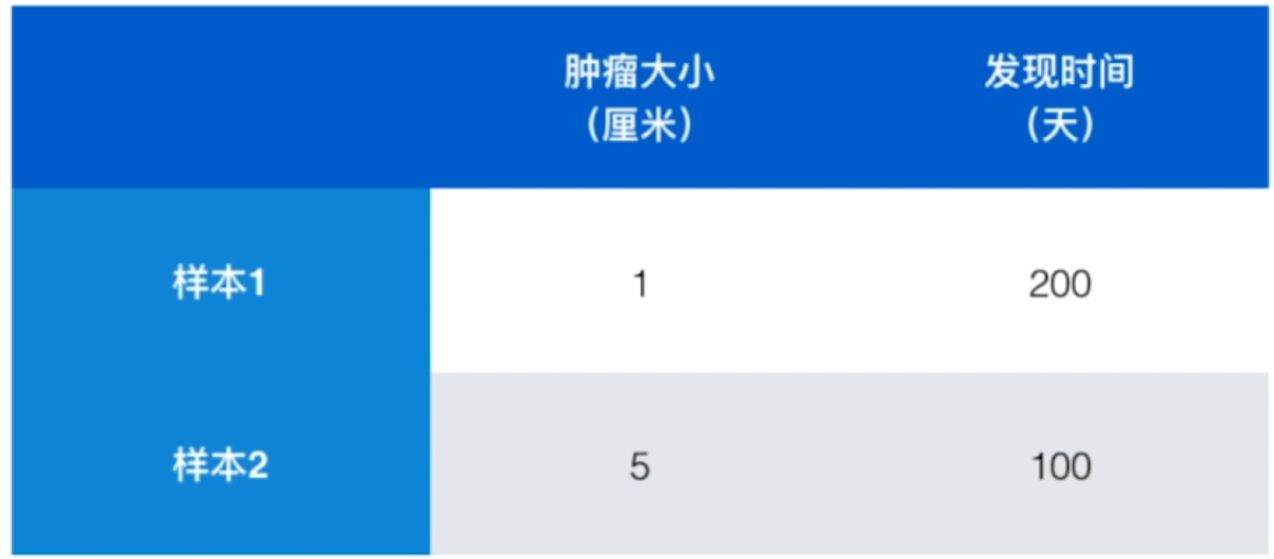
由于“发现时间”的数值是两位数,“肿瘤大小”的数值是个位数,在KNN算法进行“距离”运算后,很明显特征“发现时间”的影响比特征“肿瘤大小”更大,如何消除这种不同数值规模带来的“不公平”呢?
解决方法
最值归一化
把所有数据映射到 0-1 之间。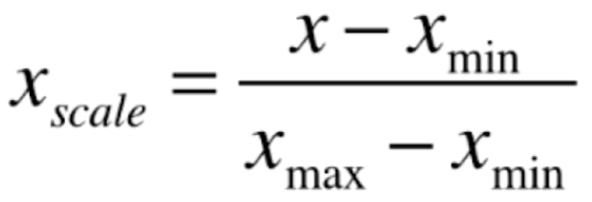
- 局限性:适用于分布有明显边界的情况。
-
均值方差归—化
[x] 算法思想:把数据平移(到坐标原点附近)、压缩(把数据)
- 原理:把所有数据归一到均值为0方差为1的分布中。
- 优点:相比最值归一化,没有了其局限性和缺点。
编码
import numpy as npimport matplotlib.pyplot as plt# 准备数据:50行 * 2列的矩阵X2 = np.random.randint(0, 100, (50, 2))X2 = np.array(X2, dtype=float)# 每列均值方差归—化X2[:,0] = (X2[:,0] - np.mean(X2[:,0])) / np.std(X2[:,0])X2[:,1] = (X2[:,1] - np.mean(X2[:,1])) / np.std(X2[:,1])# 可视化plt.scatter(X2[:,0], X2[:,1])plt.show()
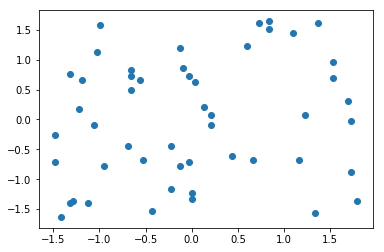
# 特征1的:均值、方差np.mean(X2[:,0]) # -1.1102230246251565e-16np.std(X2[:,0]) # 0.9999999999999999# 特征2的:均值、方差np.mean(X2[:,1]) # 1.121325254871408e-16np.std(X2[:,1]) # np.std(X2[:,1])
公式
在自然界中,大部分的数据都是符合正态分布的。
从这个角度得到启发,进行数据量纲的统一。(其实量纲统一的方式有很多)
将一个标准正态分布转化为一个普通正态分布,公式如下:
其中,为标准正态分布中的元素,
为平均值,S 为标准差。
如何理解?
- 1.将一个
标准正态分布数据样本``放大 - 2.将放大后的数据
平移
所以,生成正态分布数据时的代码如下:
data = 2000*np.random.randn (1000) +10000 # 2000为标准差,10000为均值。1000为数据样本数
将上述公式反过来,即为普通正态分布转为标准正态分布
优点
- 1.提高运算效率:
0-1范围内的数值较小,计算机计算快很多。 - 2.减小离群值的影响:数据经过压缩后在一个
0-1的范围内,对算法的影响也是微乎其微。scikit-learn中的Scaler
准备数据
```python源码
import numpy as np from sklearn import datasets
加载数据
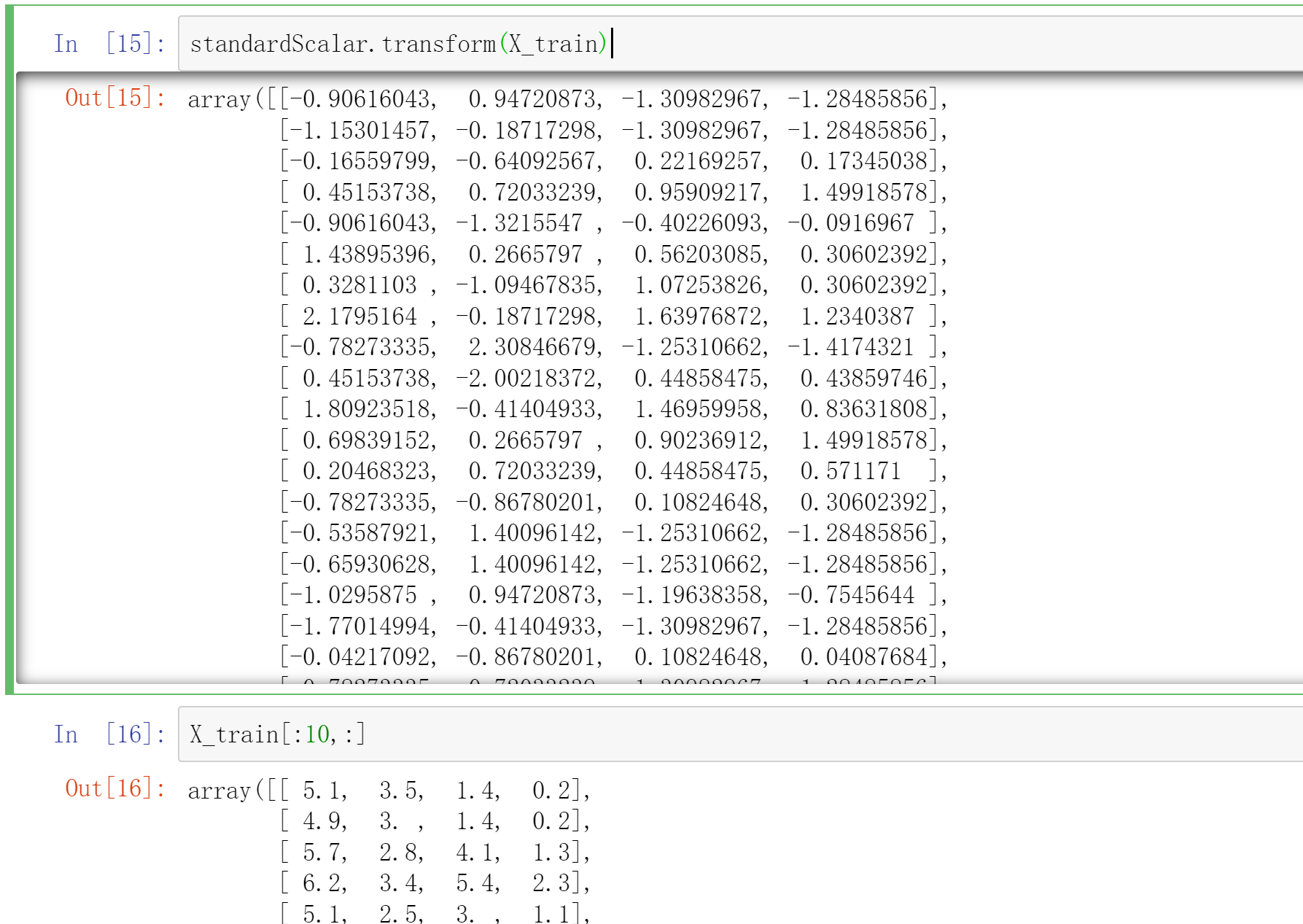
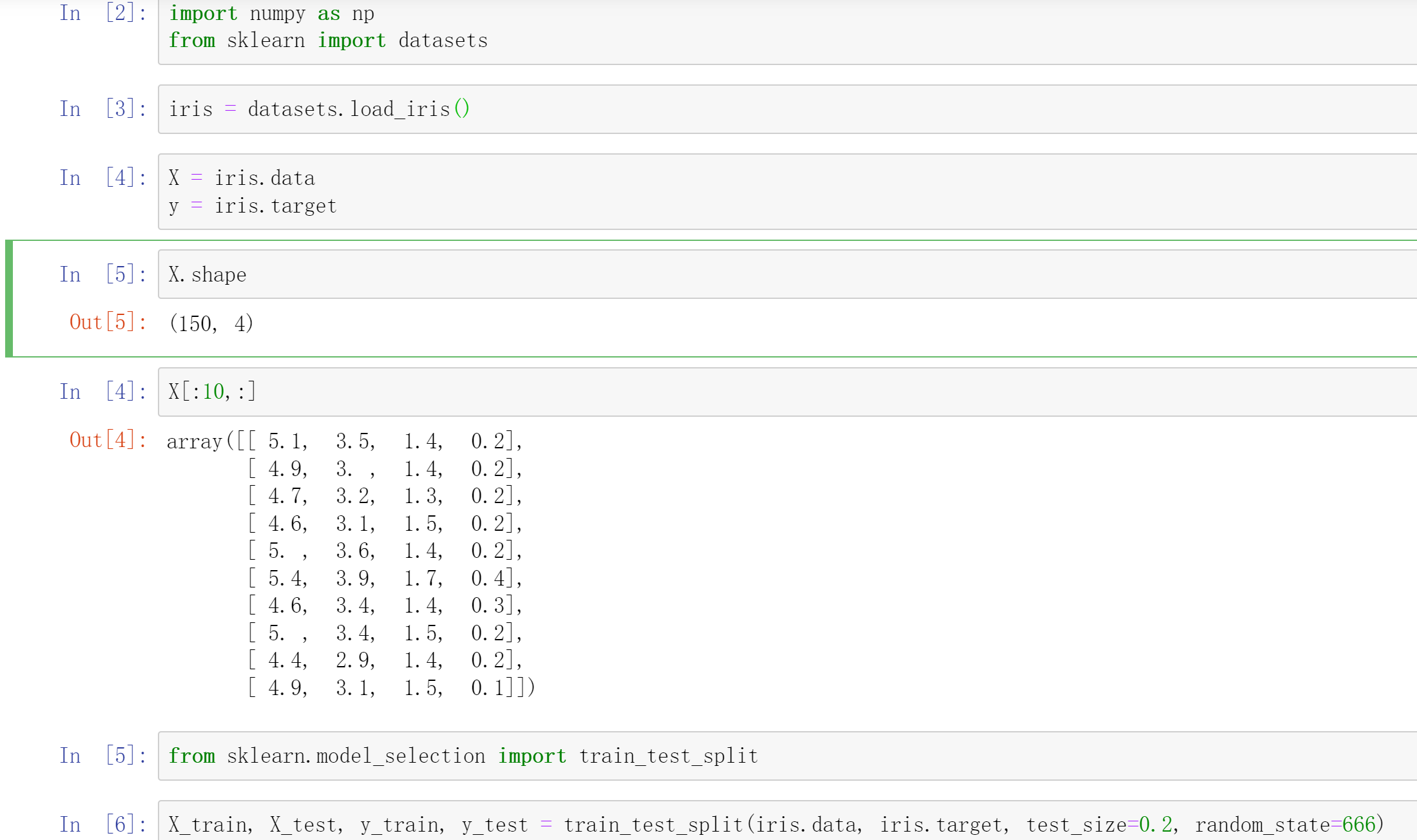
iris = datasets.load_iris() X = iris.data y = iris.target X.shape # (150, 4)
切分数据集
from sklearn.model_selection import train_test_split X_train, X_test, y_train, y_test = train_test_split(iris.data, iris.target, test_size=0.2, random_state=666)
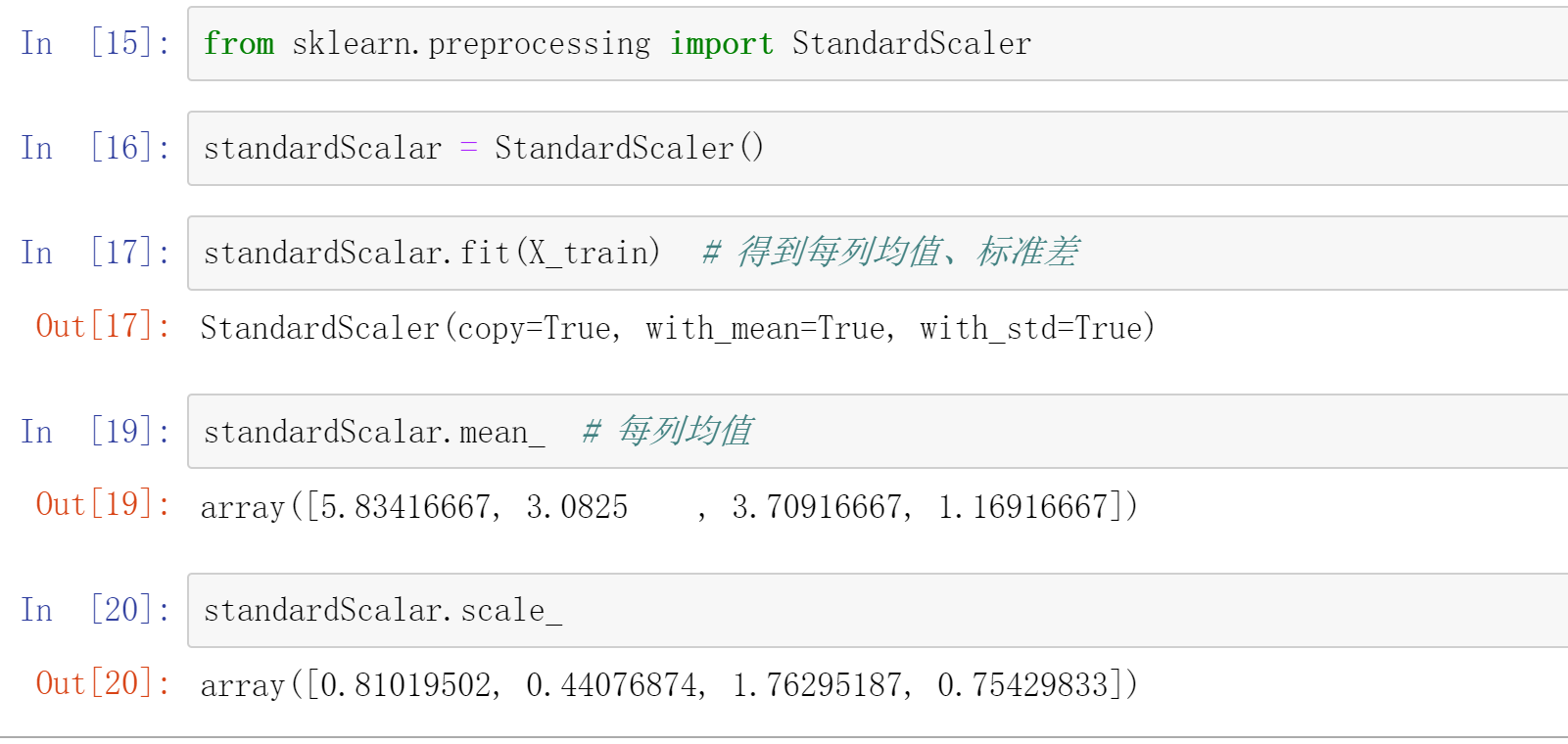
<a name="Eu4II"></a>## 归一化```python# 源码from sklearn.preprocessing import StandardScalerstandardScalar = StandardScaler()standardScalar.fit(X_train) # 得到每列均值、标准差standardScalar.mean_ # 每列均值standardScalar.scale_ # 每列标准差standardScalar.transform(X_train) # 训练集归一化X_test_standard = standardScalar.transform(X_test) # 测试集归一化
knn分类
from sklearn.neighbors import KNeighborsClassifierknn_clf = KNeighborsClassifier(n_neighbors=3)knn_clf.fit(X_train, y_train) # 建模knn_clf.score(X_test_standard, y_test) # 模型打分# 如果要预测,特征数据也要归一化

自实现standardScaler
import numpy as npclass StandardScaler:def __init__(self):self.mean_ = Noneself.scale_ = Nonedef fit(self, X):"""根据训练数据集X获得数据的均值和方差"""assert X.ndim == 2, "The dimension of X must be 2"self.mean_ = np.array([np.mean(X[:,i]) for i in range(X.shape[1])])self.scale_ = np.array([np.std(X[:,i]) for i in range(X.shape[1])])return selfdef transform(self, X):"""将X根据这个StandardScaler进行均值方差归一化处理"""assert X.ndim == 2, "The dimension of X must be 2"assert self.mean_ is not None and self.scale_ is not None, \"must fit before transform!"assert X.shape[1] == len(self.mean_), \"the feature number of X must be equal to mean_ and std_"resX = np.empty(shape=X.shape, dtype=float)for col in range(X.shape[1]):resX[:,col] = (X[:,col] - self.mean_[col]) / self.scale_[col]return resX